ک تایی
ک تایی یا k-تایی در بیوانفورماتیک، یک زیر رشته به طول k از رشته بیولوژیکی است. در ابتدا، k-تایی در ژنومیک محاسباتی و آنالیز دنباله استفاده میشد که در آن K-تایی تشکیل شده از نوکلئوتایدها (A,T، C,G) هستند. k-تاییها به خاطر بازسازی توالیهای DNA,[۱] شناسایی گونهها در نمونههای متاژنومی[۲] و تولید واکسن زنده[۳] مطرح شدند. معمولاً کلمه k-تایی به همه رشتههای دنبالهای به طول k اشاره دارد. برای مثال، رشتهٔ AGAT میتواند ۴ مونومر (A , G , A , T)، سه ۲-تایی (AG, GA, AT)، دو ۳-تایی (AGA, GAT) و یک ۴-تایی داشته باشد، که زیررشته ۴-تایی برابر خود رشتهاست. بهطور کلی یک رشته به طول L میتواند L-k+1 رشته k-تایی داشته باشد. همچنین برای n مونومر، n^k تا k-تایی ممکن وجود دارد؛ که در رشته DNA تعداد مونومرها برابر با ۴ است.
معرفی
[ویرایش]k-تاییها زیر رشتههایی به طول k هستند. برای مثال در جدول زیر تمام k-تاییها برای یک رشته نمونه از DNA را مشاهده میکنید.
| k | k-تایی |
|---|---|
| ۱ | G, T, A, G, A, G, C, T, G, T |
| ۲ | GT, TA, AG, GA, AG, GC, CT, TG, GT |
| ۳ | GTA, TAG, AGA, GAG, AGC, GCT, CTG, TGT |
| ۴ | GTAG, TAGA, AGAG, GAGC, AGCT, GCTG, CTGT |
| ۵ | GTAGA, TAGAG, AGAGC, GAGCT, AGCTG, GCTGT |
| ۶ | GTAGAG, TAGAGC, AGAGCT, GAGCTG, AGCTGT |
| ۷ | GTAGAGC, TAGAGCT, AGAGCTG, GAGCTGT |
| ۸ | GTAGAGCT, TAGAGCTG, AGAGCTGT |
| ۹ | GTAGAGCTG, TAGAGCTGT |
| ۱۰ | GTAGAGCTGT |
طیف k-تایی
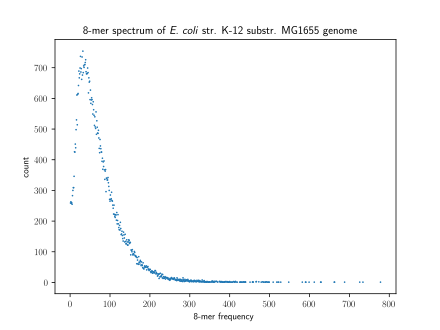
[ویرایش]یک روش برای نمایش دادن k-تاییها استفاده از طیف k-تایی است. طیف k-تایی یک نمایش گرافیکی از یک پایگاه دادهاست که نشان میدهد چند تا k-تایی به تعداد مشخصی تکرار شدهاند؛ بنابراین محور x آن فرکانس تکرار k-تایی را نشان میدهد و محور y تعداد k-تاییهایی که به آن اندازه تکرار شدهاند.[۴][۵] شکل توزیع طیف k-تایی اطلاعات مفیدی از ویژگیهای نمونه بیولوژیکی را به ما میدهد.[۶] تعداد مدها در توزیع طیف k-تایی برای ژنوم یک گونه میتواند متفاوت باشد. در این بین تکمدیها بیشترین آمار تعداد گونه را دارند. در حالیکه تمام پستانداران توزیع طیف k-تایی چندنمایی دارند. همچنین در یک طیف k-تایی، توزیع در مناطق مختلف ژنوم میتواند متفاوت باشد. برای مثال انسانها در مناطق ترجمه نشده '۵ و اگزونها طیف تکمدی دارند و در مناطق ترجمه نشده '۳ و اینترونها طیف چندنمایی دارند.
عوامل تأثیرگذار در فراوانی k-تایی DNA
[ویرایش]تعداد k-تاییها از عوامل متعددی در سطوح مختلف تأثیر میپذیرند که معمولاً با هم در تعارضاند. لازم است ذکر شود که k-تاییها با k بزرگتر از عوامل تأثیرگذار روی k-تایی با k کوچکتر تأثیر میپذیرند. برای مثال اگر مونومر A وجود نداشته باشد، هیچکدام از ۲-تاییهای شامل A هم نمیتوانند وجود داشته باشند. به این ترتیب عوامل مختلف با یکدیگر پیوند میخورند.
k = ۱
[ویرایش]در مثال بالا، در حالتی که k=۱ باشد، ۴ تا k-تایی برای DNA داریم (A,T،G,C). در سطح مولکولی ۳ پیوند هیدروژنی بین G,C وجود دارد. در حالی که در بین A,T فقط دو پیوند هیدروژنی برقرار میشود؛ بنابراین، پیوندهای بین G,C قویتر از پیوندهای بین A,T است.[۷]
پرندگان و پستانداران تعداد بیشتری G,C نسبت به A,T دارند (محتوی CG). که منجر به فرضیهای شد که ثبات حرارتی یک عامل تفاوت تعداد CGها است. هرچند فرضیه امیدوارکننده بود، این فرضیه مورد بررسی قرار نگرفت، چرا که آنالیز روی انواع پروکاریوتها هیچ شواهدی از رابطهٔ فراوانی CGها با دما نشان نداد. در حقیقت اگر انتخاب طبیعی نیروی محرکهٔ تفاوت فراوانی CGها باشد، منجر به چند ریختی تک نوکلئوتیدی میشود که معمولاً جایگزینی مترادف است و تناسب یک ارگانیسم را تغییر نمیدهد.
فرآیند تبدیل به GC ها بسیار شبیه فرآیند انتخاب طبیعی است. به فرآیند تبدیل A و T ها به G و C تبدیل بایاس GC میگویند و آن را با gBGC نشان میدهند.[۸] این فرآیند در طی فرآیند نوترکیبی ژنی رخ میدهد.[۹] و جالب است بدانید که GC ها در اعضایی که تبدیلات زیاد دارند بیشتر هستند.[۱۰] و فقط پروکاریوتها نیستند که تبدیل GC دارند. هنوز به طور قطع مشخص نیست که آیا gBGC با انتخاب طبیعی یکی هست یا اینکه کاملا فرآیند متفاوتی است و ممکن است حتی ضعف تکاملی داشته باشد. هنوز مکانیزم دقیق gBGC و مزایا و معایب آن به طور دقیق مشخص نیست.[۱۱]
k=2
[ویرایش]برخلاف تغییرات فراوانی در مونومرها که به خاطر وجود تغییرات GC بسیار گسترده بود، تغییرات دو نوکليوتیدی خیلی متداول نیستند. بایاس دو نوکلئوتیدی یعنی فراوانی نسبی دو نوکلئوتید کنار هم و به شکل تعریف میشود که در آن فراوانی دو نوکلئوتید به هم چسبیده XY است و فراوانی نوکلئوتید X و فراوانی نوکلئوتید Y به تنهایی است. شکل ارتقا یافته این فرمول به شکل نمایش داده میشود که شامل فراوانی خود رشته و عکس مکمل آن نیز میشود.[۱۲] برخلاف GCها بایاس دو نوکئوتیدی اکثرا در طول ژنوم ثابت میمانند. بایاس دو نوکلئوتیدی تحت ترجمه به وجود نمیآیند چرا که اگر چنین بود باید الگوهای متنوعی از بایاس در مناطق رمز شده و بیرمز دیانای مشاهده میشد. همچنین،مشاهده شده که دیانای ویروس بیشتر به خانواده آن ویروس شبیه است تا به سلول میزبان آن. بنابراین میتوان نتیجه گرفت که عواملی که باعث به وجود آمدن بایاس دو نوکلئوتیدی میشوند مستقل از ترجمه عمل میکنند.
مطالعات روی بیش از ۵۰ کیلو بایت کانتیگ دیانای از یک جاندار نشان داده که بایاس دو نوکلئوتیدی در کل ژنوم تقریبا یکسان است. همچنین مشخص شده که بایاس دو نوکلئوتیدیدرحالتی که کانتیگها از یک جاندار باشند بیشتر مرتبط هستند تا وقتی که کانتیگها از جدانداران مختلف باشند.[۱۲]
منابع
[ویرایش]- ↑ Compeau, Phillip E C; Pevzner, Pavel A; Tesler, Glenn (2011-11). "How to apply de Bruijn graphs to genome assembly". Nature Biotechnology (به انگلیسی). 29 (11): 987–991. doi:10.1038/nbt.2023. ISSN 1087-0156. PMC 5531759. PMID 22068540.
{{cite journal}}: Check date values in:|date=(help)نگهداری یادکرد:فرمت پارامتر PMC (link) - ↑ Beiko, Robert G.; Perry, Scott C. (2010-01-01). "Distinguishing Microbial Genome Fragments Based on Their Composition: Evolutionary and Comparative Genomic Perspectives". Genome Biology and Evolution (به انگلیسی). 2: 117–131. doi:10.1093/gbe/evq004. PMC 2839357. PMID 20333228.
{{cite journal}}: نگهداری یادکرد:فرمت پارامتر PMC (link) - ↑ Kunec, Dusan; Osterrieder, Nikolaus; Trimpert, Jakob; Eschke, Kathrin (2018-01-29). "Attenuation of a very virulent Marek's disease herpesvirus (MDV) by codon pair bias deoptimization". PLOS Pathogens (به انگلیسی). 14 (1): e1006857. doi:10.1371/journal.ppat.1006857. ISSN 1553-7374. PMC 5805365. PMID 29377958.
{{cite journal}}: نگهداری یادکرد:فرمت پارامتر PMC (link) - ↑ «طیف k-تایی».
- ↑ Clavijo, Bernardo J.; Wright, Jonathan; Kettleborough, George; Garcia Accinelli, Gonzalo; Mapleson, Daniel (2017-02-15). "KAT: a K-mer analysis toolkit to quality control NGS datasets and genome assemblies". Bioinformatics (به انگلیسی). 33 (4): 574–576. doi:10.1093/bioinformatics/btw663. ISSN 1367-4803. PMC 5408915. PMID 27797770.
{{cite journal}}: نگهداری یادکرد:فرمت پارامتر PMC (link) - ↑ Chor, Benny; Horn, David; Goldman, Nick; Levy, Yaron; Massingham, Tim (2009). "Genomic DNA k-mer spectra: models and modalities". Genome Biology. 10 (10): R108. doi:10.1186/gb-2009-10-10-r108. ISSN 1474-760X. PMC 2784323. PMID 19814784.
- ↑ Yakovchuk, P. (2006-01-30). "Base-stacking and base-pairing contributions into thermal stability of the DNA double helix". Nucleic Acids Research (به انگلیسی). 34 (2): 564–574. doi:10.1093/nar/gkj454. ISSN 0305-1048. PMID 16449200.
- ↑ Mugal, Carina F.; Weber, Claudia C.; Ellegren, Hans (2015). "GC-biased gene conversion links the recombination landscape and demography to genomic base composition". BioEssays (به انگلیسی). 37 (12): 1317–1326. doi:10.1002/bies.201500058. ISSN 1521-1878.
- ↑ Roux, Camille; Romiguier, Jonathan (2017). "Analytical Biases Associated with GC-Content in Molecular Evolution". Frontiers in Genetics (به انگلیسی). 8. doi:10.3389/fgene.2017.00016. ISSN 1664-8021. PMC 5309256. PMID 28261263.
{{cite journal}}: نگهداری یادکرد:فرمت پارامتر PMC (link) - ↑ Spencer, C. C. A. (2006-08-01). "Human polymorphism around recombination hotspots". Biochemical Society Transactions (به انگلیسی). 34 (4): 535–536. doi:10.1042/BST0340535. ISSN 0300-5127. PMID 16856853.
- ↑ "Book sources". Wikipedia (به انگلیسی). doi:10.1002/9780470015902.a0020834.pub2.
- ↑ ۱۲٫۰ ۱۲٫۱ Karlin, Samuel (1998-10-01). "Global dinucleotide signatures and analysis of genomic heterogeneity". Current Opinion in Microbiology. 1 (5): 598–610. doi:10.1016/S1369-5274(98)80095-7. ISSN 1369-5274.





