مونو نگا ویرالس
| Mononegavirales | |
|---|---|

| |
| Vesicular stomatitis virus (VSV) virion and Mononegavirales genomes | |
| طبقهبندی ویروسها | |
| Virus group: | Group V ((-)ssRNA) |
| راسته: | Mononegavirales |
| Families | |
مونونگاویروسسانان (به لاتین: Mononegavirales)، دستهای از ویروسهای آرانای رشته منفی است که دارای ژنوم غیرقطعی هستند. برخی از اعضای رایج این راسته عبارتند از: ویروس ابولا، ویروس سنسیشیال تنفسی انسانی، ویروس سرخک، ویروس اوریون، ویروس نیپا و ویروس هاری. همه این ویروسها باعث بیماری قابل توجهی در انسان میشوند. بسیاری از پاتوژنهای مهم دیگر جانوران و گیاهان غیرانسانی نیز در این گروه قرار دارند. این ترتیب شامل یازده خانواده ویروس است: Artoviridae , Bo آرانایviridae , Filoviridae , Lispiviridae , Mymonaviridae , Nyamiviridae , Paramyxoviridae , Pneumoviridae، Rhabdoviridae , Sunviridae، و XXI.[۱]
استفاده از اصطلاح
[ویرایش]راسته Mononegavirales (تلفظ: /ˌmɒnəˌnɛɡəviˈrɑːlɪz/ MON-ə-NEG-ə-vee-RAH-liz)[note ۱][۲][۳] یک تاکسون ویروسشناسی است که در سال ۱۹۹۱ ایجاد شد[۴] و در ۱۹۹۵، ۱۹۹۷،[۵] ۲۰۰۰، ۲۰۰۵، ۲۰۱۱، ۲۰۱۶،[۶] ۲۰۱۷ اصلاح شد،[۷] و 2018.[۱] نام Mononegavirales از صفت یونانی باستان μóνος monos (که اشاره به ژنوم تکجانبی و تک رشتهای اکثر تکنگاویروسها)، فعل لاتین negare (که اشاره به قطبیت منفی این ژنومها دارد) و پسوند طبقهبندی -virales گرفته شدهاست. یک سفارش ویروسی).[۳]
معیارهای درج سفارش
[ویرایش]
یک ویروس عضوی از راسته Mononegavirales است اگر[۳]
- ژنوم آن یک آرانای خطی، بهطور معمول (اما نه همیشه) بدون قطعه، تک رشتهای و غیر عفونی با قطبیت منفی است. دارای انتهای ۳' و ۵' مکمل معکوس است. و به طور کووالانسی به پروتئینی مرتبط نیست.
- ژنوم آن دارای مرتبه ژنی مشخصه 3'-UTR - ژنهای پروتئین هسته - ژنهای پروتئین پوششی - ژن آرانای پلیمراز وابسته به آرانای - 5'-UTR (3'-NPMGL-۵') است (البته استثناهایی وجود دارد).
- ۵ تا ۱۰ mآرانای متمایز از ژنوم خود را از طریق رونویسی متوالی قطبی از یک پروموتر منفرد واقع در انتهای '۳ ژنوم تولید میکند. mآرانایها ۵' پوشیده و پلی آدنیله هستند.
- با سنتز آنتی ژنوم کامل تکثیر میشود.
- ریبونوکلئوکپسیدهای مارپیچ عفونی را به عنوان الگوهایی برای سنتز mآرانایها، آنتی ژنومها و ژنومها تشکیل میدهد.
- این یک آرانای پلیمراز وابسته به آرانای (آردی آر پی، L) را کد میکند که بسیار همولوگ با سایر مونونگا ویروسها است. و/یا
- معمولاً (اما نه همیشه) ویریون پوششی با جرم مولکولی ×۱۰۶ 106 تولید میکند. یک S 20W 550–>1045; و چگالی شناور در CsCl از 1.18-1.22 g/cm 3.
چرخه زندگی
[ویرایش]
چرخه زندگی مونونگا ویروس با اتصال ویریون به گیرندههای سطح سلولی خاص آغاز میشود و به دنبال آن ادغام پوشش ویریون با غشای سلولی و انتشار همزمان نوکلئوکپسید ویروس در سیتوزول انجام میشود. ویروس آردی آر پی تا حدی نوکلئوکپسید را میپوشاند و ژنها را به mآرانایهای رشتهای مثبت رونویسی میکند، که سپس به پروتئینهای ساختاری و غیرساختاری ترجمه میشوند.
Mononegavirus آردی آر پیs به یک پروموتور منفرد که در انتهای ۳' ژنوم قرار دارد متصل میشود. رونویسی یا پس از یک ژن خاتمه مییابد یا به ژن بعدی در پایین دست ادامه مییابد. این به این معنی است که ژنهای نزدیک به انتهای ۳ ژنوم با بیشترین فراوانی رونویسی میشوند، در حالی که ژنهایی که در انتهای ۵ هستند کمترین احتمال رونویسی را دارند؛ بنابراین ترتیب ژن یک شکل ساده اما مؤثر از تنظیم رونویسی است. فراوانترین پروتئین تولید شده، نوکلئوپروتئین است که غلظت آن در سلول تعیین میکند که آردی آر پی از رونویسی ژن به همانندسازی ژنوم تغییر کند.
همانندسازی منجر به آنتی ژنومهای تمام قد و رشته مثبت میشود که به نوبه خود به نسخههای ژنوم نسل ویروس رشته منفی رونویسی میشوند. پروتئینهای ساختاری و ژنومهای تازه سنتز شده خود به خود جمع میشوند و در نزدیکی داخل غشای سلولی تجمع مییابند. ویریونها از سلول جوانه میزنند و پوشش خود را از غشای سلولی که از آن جوانه میزنند به دست میآورند. سپس ذرات نتاج بالغ سلولهای دیگر را برای تکرار چرخه آلوده میکنند.
پالئوویروسولوژی
[ویرایش]مونونگا ویروسها سابقه ای دارند که به چندین ده میلیون سال قبل بازمیگردد. فسیلهای مونونگا ویروس به شکل ژنهای مونونگا ویروس یا قطعات ژنی ادغام شده در ژنوم پستانداران کشف شدهاند. به عنوان مثال، «فسیلهای» ژن بورناویروس در ژنوم خفاشها، ماهیها، هیراکسها، کیسهداران، نخستیسانان، جوندگان، نشخوارکنندگان و فیلها شناسایی شدهاند.[۸][۹][۱۰][۱۱][۱۲] «فسیلهای» ژن فیلوویروس در ژنوم خفاشها، جوندگان، سوهان، تنرکها و کیسهداران شناسایی شدهاست.[۹][۱۰][۱۳][۱۴] یک «فسیل» ویروس Midway در ژنوم گورخرماهی یافت شد.[۹] در نهایت، فسیلهای رابدویروس در ژنوم سخت پوستان، پشهها، کنهها و گیاهان یافت شد.[۱۵][۱۰][۱۶][۱۷]
طبقهبندی
[ویرایش]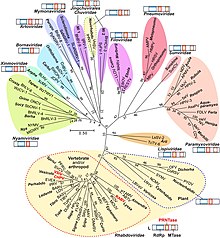
یادداشت
[ویرایش]- ↑ According to the rules for taxon naming established by the International Committee on Taxonomy of Viruses (ICTV), the name Mononegavirales is always to be capitalized, italicized, and never abbreviated. The names of the order's physical members ("mononegaviruses" or "mononegavirads") are to be written in lower case, are not italicized, and used without articles.
منابع
[ویرایش]پیوند به بیرون
[ویرایش]- ↑ ۱٫۰ ۱٫۱ Amarasinghe GK, Aréchiga Ceballos NG, Banyard AC, Basler CF, Bavari S, Bennett AJ, et al. (April 2018). "Taxonomy of the order Mononegavirales: update 2018". Archives of Virology. 163 (8): 2283–2294. doi:10.1007/s00705-018-3814-x. PMC 6076851. PMID 29637429.
- ↑ خطای یادکرد: خطای یادکرد:برچسب
<ref> غیرمجاز؛ متنی برای یادکردهای با نامKing2011وارد نشده است. (صفحهٔ راهنما را مطالعه کنید.). - ↑ ۳٫۰ ۳٫۱ ۳٫۲ Kuhn JH, Becker S, Ebihara H, Geisbert TW, Johnson KM, Kawaoka Y, et al. (December 2010). "Proposal for a revised taxonomy of the family Filoviridae: classification, names of taxa and viruses, and virus abbreviations". Archives of Virology. 155 (12): 2083–103. doi:10.1007/s00705-010-0814-x. PMC 3074192. PMID 21046175.
- ↑ Pringle, C. R. (1991). "The order Mononegavirales". Archives of Virology. 117 (1–2): 137–40. doi:10.1007/BF01310499. PMID 2006902.
- ↑ Pringle CR (1997). "The order Mononegavirales--current status". Archives of Virology. 142 (11): 2321–6. PMID 9672597.
- ↑ Afonso CL, Amarasinghe GK, Bányai K, Bào Y, Basler CF, Bavari S, et al. (August 2016). "Taxonomy of the order Mononegavirales: update 2016". Archives of Virology. 161 (8): 2351–60. doi:10.1007/s00705-016-2880-1. PMC 4947412. PMID 27216929.
- ↑ Amarasinghe GK, Bào Y, Basler CF, Bavari S, Beer M, Bejerman N, et al. (August 2017). "Taxonomy of the order Mononegavirales: update 2017". Archives of Virology. 162 (8): 2493–2504. doi:10.1007/s00705-017-3311-7. PMC 5831667. PMID 28389807.
- ↑ Horie M, Honda T, Suzuki Y, Kobayashi Y, Daito T, Oshida T, Ikuta K, Jern P, Gojobori T, Coffin JM, Tomonaga K (January 2010). "Endogenous non-retroviral RNA virus elements in mammalian genomes". Nature. 463 (7277): 84–7. Bibcode:2010Natur.463...84H. doi:10.1038/nature08695. PMC 2818285. PMID 20054395.
- ↑ ۹٫۰ ۹٫۱ ۹٫۲ Belyi VA, Levine AJ, Skalka AM (July 2010). Buchmeier MJ (ed.). "Unexpected inheritance: multiple integrations of ancient bornavirus and ebolavirus/marburgvirus sequences in vertebrate genomes". PLOS Pathogens. 6 (7): e1001030. doi:10.1371/journal.ppat.1001030. PMC 2912400. PMID 20686665.
- ↑ ۱۰٫۰ ۱۰٫۱ ۱۰٫۲ Katzourakis A, Gifford RJ (November 2010). Malik HS (ed.). "Endogenous viral elements in animal genomes". PLOS Genetics. 6 (11): e1001191. doi:10.1371/journal.pgen.1001191. PMC 2987831. PMID 21124940.
- ↑ Cui J, Wang LF (November 2015). "Genomic Mining Reveals Deep Evolutionary Relationships between Bornaviruses and Bats". Viruses. 7 (11): 5792–800. doi:10.3390/v7112906. PMC 4664979. PMID 26569285.
- ↑ Horie M, Tomonaga K (April 2018). "Paleovirology of bornaviruses: What can be learned from molecular fossils of bornaviruses". Virus Research. 262: 2–9. doi:10.1016/j.virusres.2018.04.006. PMID 29630909.
- ↑ Taylor DJ, Leach RW, Bruenn J (June 2010). "Filoviruses are ancient and integrated into mammalian genomes". BMC Evolutionary Biology. 10: 193. doi:10.1186/1471-2148-10-193. PMC 2906475. PMID 20569424.
- ↑ Taylor DJ, Dittmar K, Ballinger MJ, Bruenn JA (November 2011). "Evolutionary maintenance of filovirus-like genes in bat genomes". BMC Evolutionary Biology. 11: 336. doi:10.1186/1471-2148-11-336. PMC 3229293. PMID 22093762.
- ↑ Metegnier G, Becking T, Chebbi MA, Giraud I, Moumen B, Schaack S, Cordaux R, Gilbert C (2015). "Comparative paleovirological analysis of crustaceans identifies multiple widespread viral groups". Mobile DNA. 6: 16. doi:10.1186/s13100-015-0047-3. PMC 4573495. PMID 26388953.
- ↑ Chiba S, Kondo H, Tani A, Saisho D, Sakamoto W, Kanematsu S, Suzuki N (July 2011). Nagy PD (ed.). "Widespread endogenization of genome sequences of non-retroviral RNA viruses into plant genomes". PLOS Pathogens. 7 (7): e1002146. doi:10.1371/journal.ppat.1002146. PMC 3136472. PMID 21779172.
- ↑ Fort P, Albertini A, Van-Hua A, Berthomieu A, Roche S, Delsuc F, Pasteur N, Capy P, Gaudin Y, Weill M (January 2012). "Fossil rhabdoviral sequences integrated into arthropod genomes: ontogeny, evolution, and potential functionality". Molecular Biology and Evolution. 29 (1): 381–90. doi:10.1093/molbev/msr226. PMID 21917725.
